|
|
(21), (22) Заявка: 2005108437/13, 25.03.2005
(24) Дата начала отсчета срока действия патента:
25.03.2005
(43) Дата публикации заявки: 10.09.2006
(46) Опубликовано: 10.03.2007
(56) Список документов, цитированных в отчете о
поиске:
RU 2202178 C1, 20.04.2003. EP 0807690, 19.11.1997. SU 1611303 A1, 07.12.1990.
Адрес для переписки:
119334, Москва, ул. Вавилова, 34/5, ИБГ РАН, С.К. Семеновой
|
(72) Автор(ы):
Семенова Серафима Константиновна (RU),
Хрисанфова Галина Григорьевна (RU),
Луданный Руслан Игоревич (RU),
Рысков Алексей Петрович (RU),
Призенко Владимир Кузьмич (RU),
Богерук Андрей Кузьмич (RU)
(73) Патентообладатель(и):
ИНСТИТУТ БИОЛОГИИ ГЕНА Российской Академии Наук (ИБГ РАН, статус Государственного учреждения) (RU),
ООО “Акватехнопарк” (RU)
|
(54) БИОЛОГИЧЕСКИЙ МАРКЕР ДЛЯ ОПРЕДЕЛЕНИЯ ПОРОДЫ РЫБ, НАБОР И СПОСОБ ОПРЕДЕЛЕНИЯ ПОРОДНОЙ ПРИНАДЛЕЖНОСТИ РЫБ
(57) Реферат:
Изобретение относится к биотехнологии и рыбоводству. Предложен способ определения породной принадлежности рыб с использованием полиморфных маркеров. Способ предусматривает сбор образца крови, выделение ДНК и исследование геномной ДНК с помощью ПЦР. При осуществлении способа используют полиморфные маркеры ДНК разного размера или длины. Визуализацию ДНК проводят при электрофорезе в акриламидном или агарозном геле. Предложены также биологические маркеры и набор для осуществления такого способа. 3 н. и 3 з.п. ф-лы, 2 ил., 2 табл.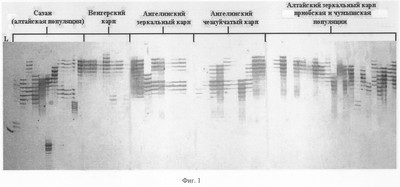
Изобретение относится к биотехнологии, в частности генетическим исследованиям, к области, связанной с породной идентификацией рыб и их гибридов, а также для прижизненного генетического мониторинга природных и искусственных популяций рыб путем анализа полиморфизма дезоксирибонуклеиновой кислоты.
Известен способ идентификации рыб и их гибридов, в котором в качестве экстракта белков используют слизь, взятую путем соскоба с наружной поверхности тела, и используют гели из полиакриламида, содержащего 4-метилумбеллиферилацетат, а электрофорез и сканирование проводят одновременно (а.с. СССР №1611303, БИ №45, 1990).
Известен способ определения родства живых организмов, включающий выделение ДНК, обработку рестрикционными эндонуклеазами, электрофоретическое фракционирование фрагментов ДНК, перенос ДНК из геля, молекулярную гибридизацию ДНК из живого организма (а.с. СССР №1552642, 1994).
Известен способ селективной амплификации ДНК, олигонуклеотид и набор для селективной амплификации (Патент РФ №2182176, 1992).
Недостатком известных способов является непригодность их для породной идентификации рыб.
Наиболее близким к предлагаемому способу определения пород карповых рыб и биологического маркера для их характеристики является способ подбора особей русского осетра ACIPENCER guldensatdti в аквакультуре, включающий свенс-анализ митохондриальной ДНК в области D-петли методом ПЦР-амплификации, в которой в процессе анализа используется прямой (OS1) и обратный (OS2) праймеры, а для воспроизводства подбирают особей, различающихся по длине ПЦР-продуктов мтДНК (патент РФ №2202178, 2001).
Недостатками этого известного способа является невозможность определения породной принадлежности рыб, отсутствие праймеров и набора для идентификации и паспортизации карповых рыб, невозможность создания генетического паспорта породы рыб.
Задачей предлагаемого изобретения является определение породной принадлежности и установление происхождения распространенных и практически значимых одомашненных форм различных рыб, в том числе из семейства карповых (Cyprinidae).
В результате использования предлагаемого биологического маркера для определения породы рыб, набора и способа идентификации и паспортизации пород рыб появляется возможность создания генетического паспорта породы и повышение эффективности идентификации в генетико-селекционных мероприятиях, связанных с разведением рыб.
Вышеуказанный результат достигается тем, что биологический маркер, содержащий полиморфную ДНК и характеризующий породную принадлежность, происхождение распространенных и практически значимых природных и одомашненных форм рыб семейства карповых и представляющий собой нуклеотидную последовательность из группы 5 пар праймеров следующего состава
| PR-4 |
F TCCAAGTCAGTTTAATCACCG |
| R GGGAAGCGTTGACAACAAGC |
| PR-10 |
F CTGCAGGGTGCAGGAATAGAC |
| R GGCTGAACAGGAACAAGAGGC |
| PR-19 |
F GAATCCTCCCCATCATGCAAAC |
| R GCACAAACTCCACATTGTGCC |
| PR-24 |
F GCTCCAGATTGCACATTATAG |
| R CTACACACACGCAGAGCCTTTC |
| PR-26 |
F CCCTGAGATAGAAACCACTG |
| R CACCATGCTTGGATGCAAAAG |
Результат достигается также тем, что используют набор для определения породной принадлежности рыб, содержащий биологический маркер, реакционную смесь, состоящую из 60 мМ трис-HCl, 10 мМ сульфата аммония, 0,1% TWEEN 20, по 100 мкМ каждого dNTP, 0,5 мкМ MgCl2, 0,9 единиц Taq-полимеразы и эталонные ДНК для каждой породы.
В способе определения породной принадлежности рыб семейства карповых, включающем сбор образца крови, выделение ДНК из него, исследование геномной ДНК с помощью ПЦР, ПЦР-анализ проводят используя предлагаемый набор, получают полиморфные маркеры ДНК разного размера, визуализируют эти маркерные ДНК с помощью электрофореза в акриламидном или агарозном геле, затем для типирования выявленных в изученной группе аллелей проводят сравнение генетического разнообразия определяемой и контрольной группы образцов амплифицируемой ДНК и по отсутствию или присутствию в исследуемой группе определенных единичных породоспецифических аллелей или аллельных сочетаний определяют породную принадлежность.
Таблица 1.
Распределение полиморфных маркеров, выявляемое в исследованных породах карпа и сазана при использовании 5 пар праймеров |
| Порода |
Аллели локуса PRL 4 |
Аллели локуса PRL 24 |
Аллели локуса PRL 19 |
Аллели локуса PRL 26 |
Аллели локуса PRL 10 |
| Амурский сазан |
1, 2, 7, 9, 10, 11, 12, 14*, |
1, 2, 5, 11, 9*, 14, 15, 16*. |
1, 2, 3, 4, 5, 7, 8, 13*, 14*. |
3, 4, 5, 8, 9, 11*, 13*, 14*. |
1, 2, 3. |
| Алтайский сазан |
1, 7, 8, 9, 10, 12, 13* |
3, 5, 7, 8, 10, 11, 14*, 15*, 17*, 18*. |
1, 2, 3, 4, 6, 7, 8. |
1, 3, 4, 5, 6, 8, 9*, 11*, 12*, 13*, 14*. |
1, 2, 4, 5, 6, 9*, 10*, 11*. |
| Ангелинский карп |
3, 4, 5, 7, 11, 15* |
5, 7, 10, 11, 12*, 15*. |
1, 2, 3, 5, 8, 12, 16*, 17*. |
1*, 2*, 3, 4, 5, 6, 8, 9, 11. |
1, 2, 4, 6, 7, 8, 11. |
| Алтайский карп (приобская и чумышская популяция) |
2, 3, 4, 7, 8, 10, 11, 16*. |
5, 6, 7, 10*, 11, 15*, 20*. |
1, 3, 5, 6*, 7*, 11*, 14*, 17*. |
1, 3, 4, 5, 6, 7, 8, 9, 10. |
1, 4, 8, 13*, 14*. |
| Венгерский карп |
2, 3, 7, 10 |
4, 5, 9 |
2, 8, 9*, 12*, 17. |
3, 4, 5, 6, 9, 10. |
1, 4, 6, 12*. |
| Ставропольский карп |
1, 4, 7, 10, 17. |
3, 5, 6, 10, 11, 14, 15. |
2, 3, 10, 13. |
3, 4, 5, 6, 8*, 10*, 13*, 14*. |
1, 2, 4. |
| Ропшинский карп |
1, 6, 9, 12, 18*. |
3, 5, 9, 10, 11, 12, 13*, 20*. |
1, 2, 3, 8*, 10*, 13. |
3, 4, 5, 6, 7, 9, 10, 11, 12. |
1, 2, 3, 4, 5, 6. |
| Черепецкий рамчатый карп |
1, 3, 5, 19* |
6*, 7*, 9*, 10*. |
1*, 9*, 13*. |
1, 2, 3, 4, 5, 7, 9*. |
1, 4, 7, 8. |
| Черепецкий чешуйчатый карп |
2, 7, 10, 20* |
1, 2, 3, 5, 7. |
3, 7, 10, 15*. |
1, 3, 4, 7. |
1, 4, 6, 7, 8. |
| Всего аллелей |
20 |
20 |
17 |
14 |
14 |
| * единичные породоспецифичные аллели или набор специфичных аллелей. |
При этом для каждой породы и породной группы карповых рыб составляют генетический паспорт, представленный в таблице 1, в которой указано распределение 50-75 полиморфных микросателлитных маркеров, выявленных с помощью 5 пар праймеров (PR 4, 10, 19, 24, 26) по пяти соответствующим локусам (PRL 4, 10, 19, 24, 26), и по которой определяют породу рыб.
Размер амплифицируемых продуктов определяют с помощью маркера молекулярных масс (10 bp Ladder).
Набор контрольных амплификатов содержит образцы амплифицированной ДНК, характерные для каждой из изученных пород карповых рыб, в том числе карпа или сазана.
В способе верификацию породной принадлежности проверяют с помощью многомерного анализа (например, дискриминантного анализа, метода главных компонент и т.п.), в котором в качестве обучающей выборки служит выборка аллелей и аллельных сочетаний одной из набора пород рыб.
Способ генетической идентификации пород рыб, в том числе рыб семейства карповых, в том числе пород карпа и сазана с применением предлагаемых биологических маркеров и набора осуществляют следующим образом.
Осуществляют геномный фингерпринтинг для выявления геномной вариабельности рыб и для идентификации и паспортизации различных пород рыб с использованием метода полимеразной цепной реакции (ПЦР или PCR). В предлагаемом способе получают молекулярно-генетические характеристики пород рыб, в том числе карповых рыб, в том числе наиболее распространенных на территории России пород карпа и сазана, предлагают породоспецифичные маркеры с целью паспортизации пород.
При изучении пород и породных групп рыб выявляют несколько породоспецифичных маркеров, которые в дальнейшем используют в качестве эталона для определения породной принадлежности любой группы рыб. Для этого собирают образцы различных тканей (кровь, слизь, кусочки плавников, мышцы и т.п.), выделяют из них ДНК, проводят амплификацию определенных участков ДНК с определенными праймерами, визуализируют полученную изменчивость с помощью специального электрофоретического разделения амплификатов и специального окрашивания, проводят типирование полученной изменчивости путем сравнения с эталонными образцами.
На заключительном этапе для каждой идентифицируемой группы составляют таблицу-паспорт с указанием распределения 50-70 аллельных вариантов по 5 полиморфным локусам. На основании этой таблицы с высокой вероятностью либо визуально, либо с помощью многомерного статистического анализа определяют принадлежность (или отсутствие принадлежности) исследуемой группы рыб к одной из наиболее распространенных пород.
Для генотипирования породы собирают образцы тканей у 20-30 представителей породы (лучше образцы крови) по следующей методике отбора проб.
Наилучшим материалом для последующего выделения суммарной ДНК являются: кровь (обычно, из хвостовой вены) и фрагменты плавников (грудных, брюшных, анальных или спинного), которые берут прижизненно, стараясь не наносить излишних повреждений донорам биопсийного материала. Используют также половые продукты рыб (овулировавшие яйцеклетки и сперму производителей), фрагменты ястыков и семенников на различных стадиях созревания. В тех случаях, когда существует возможность получения проб от умертвленных «ex tempora» особей, в качестве материала может дополнительно использоваться мышечная ткань, печень и другие внутренние органы, за исключением кишечника.
Для кодирования наиболее часто используемых видов тканей предлагается использовать систему, принятую в Российской Национальной Коллекции эталонных генетических материалов (РНКЭГМ), которая демонстрируется табл.2.
| Таблица 2. |
| |
Жабры – отдельные жаберные дуги, соскобы жаберных тычинок |
| |
Икра – неоплодотворенные овулировавшие или пробойные икринки |
| |
Ястык – женские гонады на различных стадиях созревания ооцитов до естественной или гормониндуцированной овуляции |
| |
Сперма – текучая семенная жидкость, содержащая зрелые сперматозоиды |
| |
Молоки – мужские гонады на различных стадиях сперматогенеза до периода созревания |
| |
Плавники – любые, включая хвостовой и жировой. Их фрагменты |
| |
Мышцы – любые (красные, белые) скелетные мышцы, мышечная ткань висцерального черепа |
| |
Кровь – периферическая (из хвостовой вены), при забое возможно взятие из сердца, аорты |
| |
Печень – фрагменты (у маломерных особей целиком с обязательным исключением желчного пузыря) |
| 0 |
Почки – проксимальные отделы |
| 1 |
Селезенка – целиком или частично (крупные особи) |
| 2 |
Мозг – все содержимое нейрокраниума за исключением спинного мозга |
| 3 |
Мотальный препарат – эмбрионы, постэмбрионы, личинки, ранняя молодь. Зафиксированные целиком |
| 4 |
Другое – сердце, глаза, усики, хрящевые, костные ткани, спилы и т.д. |
Консервация (фиксация) проб.
Для предотвращения деградации ДНК, полученные пробы помещают в фиксирующий раствор. Как правило, это – 70-96% этиловый спирт (этиловый ректификованный, высшей очистки).
Мышцы, фрагменты плавников, печени, ястыков и семенников перед этанольной фиксацией ножницами, скальпелем или полулезвием безопасной бритвы измельчают до кусочков, размеры которых соответствуют приблизительно 0,5 см3.
Первичную фиксацию осуществляют при соотношении спирт/образец 10/1. Вторичную – в среднем, через трое-четверо суток в соотношении 5/1.
Кровь собирают из хвостовой вены при отрезании хвостового плавника (или шприцом). Капли крови собирают в стерильную пробирку с консервантом (0,5 М ЭДТА, рН 8,0) соотношение крови и консерванта 1:10. Не допускается попадания в пробирки слизи и других посторонних примесей, способствующих образованию в пробирке сгустков.
Образцы, зафиксированные и перезалитые этанолом, могут храниться при комнатной температуре в течение 30 и более суток. Для более продолжительного хранения их следует поместить в свежий спиртовой раствор (не ниже 92%) в соотношении спирт/образец 5/1.
Образцы крови, зафиксированные 0,5 М ЭДТА, хранят в холодильнике в течение 30 суток при низких плюсовых (2-4°С) температурах, но образец не должен подвергаться замораживанию.
После замораживания все образцы хранят в холодильнике при отрицательных (не выше -50°С) температурах без размораживания. Но не дольше 90 суток. Для более продолжительного хранения пригодны кельвинаторы, поддерживающие температуру ниже -50°С, а также жидкоазотные сосуды Дюара.
Выделение и хранение ДНК.
1. Выделение ДНК из крови.
Образцы свежей крови, законсервированные в 0,05 М растворе ЭДТА (рН 8.0), хранят при температуре +4°С. Выделение ядерной ДНК проводили по методу Мэтью (Mathew, 1984). Выделение ДНК заключается в последовательном прохождении следующих фаз: лизис клеток и ядер, депротеинизация ДНК с помощью протеиназы К, фенол-хлороформная депротеинизация, осаждение и растворение ДНК. Для получения более очищенных образцов ДНК проводят дополнительную очистку с помощью набора реагентов DIAtom Prep 200 (Москва), основанного на использовании лизирующего реагента с гуанидинтиоционатом, в присутствии которого ДНК сорбируется на сорбенте и отмывается затем раствором Экстра-Гена Prep 200 (Москва), основанного на использовании лизирующего реагента с гуанидинтиоционатом, в присутствии которого ДНК сорбируется на сорбенте и отмывается затем раствором Экстра-Гена . .
2. Лизис клеток.
К образцу крови добавляют лизирующий буфер в соотношении 1 объем крови: 30 объемов буфера. Состав лизирующего буфера: 320 мМ сахарозы, 1% тритон Х-100, 5 мМ MgCl2 и 10 мМ трис-HCl (рН 7,6). Смесь инкубируют 1 час 30 минут, затем центрифугируют 15 минут при 4°С (2000 g). Супернатант сливают, а осадок подсушивают в течение 30 минут при комнатной температуре.
3. Суспендирование и лизис ядер.
К полученному осадку добавляют 4 мл буфера (25 мМ ЭДТА и 75 М NaCl). Затем проводят суспендирование ядер резким встряхиванием пробирки в течение 10 минут. К суспензии добавляют 0,4 мл 10% SDS (рН 8,0) и выдерживают в течение 10 минут, периодически плавно перемешивая. По завершению лизиса ядер к суспензии добавляют по каплям протеиназу К до конечной концентрации 50 мкг/мл, осторожно перемешивают и инкубируют смесь 4 часа при 45°С или оставляют на ночь при 37°С.
4. Депротеинизация ДНК.
В пробирку со смесью, содержащей лизированные ядра, добавляют 0,3 мл 5 М ацетата натрия или 0,3 мл 5 М ацетата аммония, или 0,5 мл 3 М перхлората натрия и аккуратно перемешивают. К смеси добавляют равный объем фенола, насыщенного трис-HCl (рН>7,6), плавно перемешивают 5 минут и приливают 5 мл водонасыщенного хлороформа, снова плавно перемешивают 5 минут и центрифугируют при 4°С в течение 20 минут (2000 g). Верхнюю фазу с растворенной ДНК отбирают пипеткой, стараясь не захватывать интерфазу, содержащую белки. Фенол-хлороформная депротеинизация повторяется 2-3 раза до полной очистки раствора ДНК от белков. На следующей стадии к отобранному супернатанту добавляют 10 мл водонасыщенного хлороформа, плавно перемешивают 5 минут и центрифугируют в течение 10 минут (2000 g) и отбирают верхнюю фазу с растворенной в ней ДНК.
5. Осаждение ДНК.
К 1 мл растворенной ДНК приливают 20 мкл 5 М ацетата натрия и осторожно перемешивают. Затем добавляют тройной объем 96° этанола и плавно перемешивают до выпадения ДНК в виде тонких белых нитей или белого осадка. ДНК однократно промывают в 70° этаноле для удаления избытка соли, затем – в 90° этаноле. На следующем этапе спирт сливают, ДНК высушивают на воздухе до полного испарения спирта. Высушенную ДНК растворяют в 100 мкл дистиллированной воды.
6. Дополнительная очистка ДНК
В случае некачественного выделения ДНК из крови препараты ДНК необходимо дополнительно очистить. Один из методов дополнительной очистки состоит в следующем: к 40 мкл растворенной ДНК добавляют 160 мкл лизирующего агента и перемешивают содержимое. После термостатирования в течение 5-7 минут при температуре 65°С смесь центрифугируют и супернатант переносят в чистую пробирку. Далее в супернатант добавляют 8 мкл суспензии сорбента NucleoS , пробирку помещают на ротатор и перемешивают 10 мин. Смесь центрифугируют 10 с при 5000 g, затем с помощью водоструйного насоса удаляют супернатант, не задевая осадка. К осадку добавляют 80 мкл лизирующего реагента, перемешивают до гомогенного состояния, добавляют 200 мкл солевого буфера и центрифугируют после перемешивания 10 с при 5000 g. Затем удаляют супернатант с помощью водоструйного насоса, не задевая осадка, и добавляют 200 мкл солевого буфера, перемешивают и центрифугируют 10 с при 5000 g. Операцию повторяют три раза. Далее пробирки с осадком высушивают при температуре 65°С 4-5 мин, добавляют 20 мкл раствора Экстрагена, суспендируют содержимое пробирки на вортексе 5-10 с и термостатируют 4-5 мин при 65°С. Затем еще раз суспендируют на вортексе и центрифугируют 1 мин при 10000 g. Супернатант, содержащий очищенную ДНК, переносят в чистую пробирку и хранят при температуре +4°С. , пробирку помещают на ротатор и перемешивают 10 мин. Смесь центрифугируют 10 с при 5000 g, затем с помощью водоструйного насоса удаляют супернатант, не задевая осадка. К осадку добавляют 80 мкл лизирующего реагента, перемешивают до гомогенного состояния, добавляют 200 мкл солевого буфера и центрифугируют после перемешивания 10 с при 5000 g. Затем удаляют супернатант с помощью водоструйного насоса, не задевая осадка, и добавляют 200 мкл солевого буфера, перемешивают и центрифугируют 10 с при 5000 g. Операцию повторяют три раза. Далее пробирки с осадком высушивают при температуре 65°С 4-5 мин, добавляют 20 мкл раствора Экстрагена, суспендируют содержимое пробирки на вортексе 5-10 с и термостатируют 4-5 мин при 65°С. Затем еще раз суспендируют на вортексе и центрифугируют 1 мин при 10000 g. Супернатант, содержащий очищенную ДНК, переносят в чистую пробирку и хранят при температуре +4°С.
В отдельных случаях для быстроты можно использовать различные наборы для выделения ДНК. Ниже приводим метод выделения ДНК из различных тканей рыб с помощью одного из таких наборов, а именно набора реагентов Diatom DNA Ргер200 (Москва). DNA Ргер200 (Москва).
Набор реагентов Diatom DNA Ргер200 основан на использовании Лизирующего реагента с гуанидинтиоционатом, который предназначен для лизиса клеток, солюбилизации клеточного дебриса, а также для денатурации клеточных нуклеаз. В присутствии Лизирующего реагента ДНК эффективно сорбируется на NucleoS DNA Ргер200 основан на использовании Лизирующего реагента с гуанидинтиоционатом, который предназначен для лизиса клеток, солюбилизации клеточного дебриса, а также для денатурации клеточных нуклеаз. В присутствии Лизирующего реагента ДНК эффективно сорбируется на NucleoS -сорбенте, затем легко отмывается от белков и солей спиртовым раствором. ДНК, элюированная из сорбента раствором ЭкстраГеном Е -сорбенте, затем легко отмывается от белков и солей спиртовым раствором. ДНК, элюированная из сорбента раствором ЭкстраГеном Е или чистой водой, может напрямую использоваться по назначению. или чистой водой, может напрямую использоваться по назначению.
Продолжительность выделения ДНК из жидких проб составляет 25 минут. Выделение ДНК из сухих пятен или мелкоизмельченного твердого материала может длиться до нескольких часов в зависимости от обрабатываемого материала.
7. Аналитический электрофорез
Для определения концентрации и качества выделенных образцов используют метод электрофореза в агарозном геле. Гель-электрофорез проводится в горизонтальной камере в 1х трис-боратном буфере (ТВЕ), рН 7,5-7,8 (0,089 М трис-бората, 0,089 М борной кислоты и 0,002 М ЭДТА). При внесении ДНК в карманы геля используется буфер для нанесения (ксиленцианол, бромфеноловый синий и глицерин). После электрофореза гель окрашивают в растворе бромистого этидия в течение 5-10 минут или добавляют бромистый этидий непосредственно в гель. Вхождение ДНК в гель осуществляют при 20 V в течение 15-20 минут, далее электрофорез ведут при 60 V в течение 2-3 часов. В качестве маркера концентрации используется ДНК фага  , внесенная в разных количествах, например 0,5; 1; 2 мкг. , внесенная в разных количествах, например 0,5; 1; 2 мкг.
Полимеразная цепная реакция (PCR).
1. Проведение PCR.
Выделенную ДНК используют в качестве матрицы в реакции амплификации.
Амплификацию с единичными праймерами проводят по стандартной методике на термоциклере «MJ Research» (USA), имеющем металлический нагревательный блок на 60 образцов. Реакционная смесь объемом 25 мкл содержит 60 мМ трис-HCl, 10 мМ сульфата аммония, 0,1% TWEEN 20, по 100 мкМ каждого dNTP, 0,5 мкМ MgCl2, 0,1 мкМ каждого праймера, 0,9 единиц Taq-полимеразы и 25-100 нг тотальной ДНК. Для проведения PCR используют пять пар праймеров следующего состава:
| PR-4 |
F TCCAAGTCAGTTTAATCACCG |
| R GGGAAGCGTTGACAACAAGC |
| PR-10 |
F CTGCAGGGTGCAGGAATAGAC |
| R GGCTGAACAGGAACAAGAGGC |
| PR-19 |
F GAATCCTCCCCATCATGCAAAC |
| R GCACAAACTCCACATTGTGCC |
| PR-24 |
F GCTCCAGATTGCACATTATAG |
| R CTACACACACGCAGAGCCTTTC |
| PR-26 |
F CCCTGAGATAGAAACCACTG |
| R CACCATGCTTGGATGCAAAAG |
Для предотвращения испарения с поверхности раствора во время амплификации в каждую пробирку добавляют каплю (около 15 мкл) минерального масла. Каждый цикл полимеразной реакции состоит из следующих этапов денатурации (94°С, 2 мин), отжига (36°С, 1 мин) и элонгации (72°С, 2 мин). Циклы повторяются 35 раз. Первый цикл предваряли денатурацией в течение 5 минут при температуре 94°С. После окончательной достройки амплифицированной ДНК (72°С, 2 мин) температуру снижают до 4°С.
2. Электрофоретическое разделение продуктов и визуализация ПЦР-продуктов.
Продукты амплификации подвергают аналитическому электрофорезу в 1.5% агарозном геле толщиной 7-10 мм, содержащем бромистый этидий. В качестве маркеров молекулярного веса используется 50 bp Ladder (Fermentas). Вхождение в гель осуществляют при 20 V в течение 20 минут, а разделение фрагментов – в течение 10-20 часов при 40-50 V.
В случае эффективной амплификации продукты амплификации подвергают электрофоретическому разделению в вертикальной камере в 6% ПААГе, содержащем 7% мочевину.
Визуализацию осуществляют путем окрашивания гелей азотнокислым серебром из набора Silver Sequence Staining Kit [Promega, USA] согласно инструкции производителя, с последующим фотографированием цифровым фотоаппаратом Nicon. Размер амплифицируемых продуктов определяют с помощью маркера молекулярных масс (50 bp Ladder).
Затем проводят статистическую обработку результатов и составляют породный генетический паспорт.
Для типирования (определения размера в п.н.) выявленных в изученной группе рыб аллелей необходимо провести обязательное сравнение генетического разнообразия данной и контрольной (эталонной) группы образцов амплифицируемой ДНК. Набор контрольных амплификатов может содержать от 5 до 20 образцов амплификатов, характерных для 5-20 микросателлитных аллелей. Отсутствие или присутствие в исследуемой группе определенных аллелей или аллельных сочетаний позволяет надежно определять породную принадлежность. Верификацию породной принадлежности проводят с помощью различных вариантов многомерного (дискриминантного анализа или метода главных компонент), в котором в качестве обучающей выборки может служить выборка аллелей и аллельных сочетаний одной из 9 ранее изученных пород карпа и сазана. Список пород и породных групп карпа и сазана, которые могут служить обучающей выборкой и для которых известно генетическое разнообразие и породоспецифичные аллели: карп зеркальный и чешуйчатый Ангелинский, карп Венгерский, карп Ропшинский, Черепецкий рамчатый и чешуйчатый карп, ставропольский карп, Алтайский и Амурский сазан.
На фиг.1 представлен набор полиморфных ДНК, выявляемых с помощью праймера PR-10 у различных пород карпа.
На фиг.2 представлен набор полиморфных ДНК, выявляемых с помощью праймера PR-24 у различных пород карпа.
Примеры выполнения предлагаемого способа.
Пример 1. Способ определения породы карпа с помощью праймера PR-10
Из образцов крови, полученных из особей Венгерского карпа, Алтайского карпа, Ангелинского карпа (зеркального и чешуйчатого) и Алтайского сазана, выделяют ДНК способом, указанным выше. На каждой из выделенных ДНК осуществляют ПЦР с одной из пяти пар предлагаемых праймеров, а именно PR-10. Полученные амплификационные продукты визуализируют путем электрофореза в гелях, и после окрашивания гелей серебром идентифицируют по размеру (длине) выявляемые полиморфные маркеры. На фиг.1 представлены полиморфные маркеры для каждой породы Венгерского карпа (дорожки 11-17), Алтайского карпа (дорожки 36-56), Ангелинского зеркального (дорожки 18-25) и чешуйчатого (дорожки 26-35) карпа и Алтайского сазана (дорожки 1-10).
Каждая из этих пород характеризуется определенным набором или сочетанием аллелей, часть из которых представлена в таблице 1. Это распределение служит эталоном при сравнении и идентификации неизвестных пород карповых рыб.
Путем сравнения распределения полиморфных ДНК разной длины среди исследуемых и эталонных образцов определяют породную принадлежность.
Пример 2. Способ определения породы карпа с помощью праймера PR-24.
Из образцов крови, полученных из Чумышской и Приобской популяции Алтайского карпа, Ангелинского чешуйчатого карпа и Ангелинского зеркального карпа, Амурского сазана, выделяют ДНК способом, указанным выше. На каждый из выделенных ДНК осуществляют ПЦР с одной из пяти пар предлагаемых праймеров, а именно PR -10. Полученные амплификационные продукты визуализируют путем электрофореза в гелях, и после окрашивания гелей серебром идентифицируют по размеру (длине) выявляемые полиморфные маркеры. На фиг.2 представлены полиморфные маркеры для каждой породы Чумышской (дорожки 1-10) и Приобской (дорожки 11-20), популяции Алтайского карпа, Ангелинского чешуйчатого карпа (дорожки 21-30), и Ангелинского зеркального карпа (дорожки 31-40), Амурского сазана (дорожки 41-49).
Каждая из этих пород характеризуется определенным набором или сочетанием аллелей, часть из которых представлена в таблице 1. Это распределение служит эталоном при сравнении и идентификации неизвестных пород карповых рыб.
Путем сравнения распределения полиморфных ДНК разной длины среди исследуемых и эталонных образцов определяют породную принадлежность.
Генетический паспорт составляется для каждой известной породы и породной группы и представлен в таблице 1, в которой указано распределение 50-70 полиморфных маркеров по 5 парам праймеров. Аналогичный паспорт можно составить для любой выборки карпа неизвестного происхождения.
Формула изобретения
1. Биологический маркер, содержащий полиморфную ДНК и характеризующий породную принадлежность, происхождение распространенных и практически значимых природных и одомашненных форм рыб семейства карповых и представляющий собой нуклеотидную последовательность из группы 5 пар праймеров следующего состава:
| PR-4 |
F TCCAAGTCAGTTTAATCACCG |
| R GGGAAGCGTTGACAACAAGC |
| PR-10 |
F CTGCAGGGTGCAGGAATAGAC |
| R GGCTGAACAGGAACAAGAGGC |
| PR-19 |
F GAATCCTCCCCATCATGCAAAC |
| R GCACAAACTCCACATTGTGCC |
| PR-24 |
F GCTCCAGATTGCACATTATAG |
| R CTACACACACGCAGAGCCTTTC |
| PR-26 |
F CCCTGAGATAGAAACCACTG |
| R CACCATGCTTGGATGCAAAAG |
2. Набор для определения породной принадлежности рыб семейства карповых, содержащий биологический маркер по п.1, реакционную смесь, состоящую из 60 мМ трис-HCl, 10 мМ сульфата аммония, 0,1% TWEEN 20, по 100 мкМ каждого dNTP, 0,5 мкМ MgCl2, 0,9 единиц Taq-полимеразы и эталонные ДНК для каждой породы.
3. Способ паспортизации пород рыб семейства карповых с использованием полиморфных маркеров, включающий сбор образца крови, выделение ДНК из него, исследование геномной ДНК с помощью ПЦР, отличающийся тем, что ПЦР-анализ проводят, используя набор по п.2, получают полиморфные маркеры ДНК разного размера или длины, визуализируют эти ДНК при электрофорезе в акриламидном или агарозном геле, затем для типирования выявленных в изученной группе аллелей проводят сравнение генетического разнообразия определяемой и контрольной группы образцов амплифицируемой ДНК, и по отсутствию или присутствию в исследуемой группе определенных единичных породоспецифических аллелей или аллельных сочетаний определяют породную принадлежность, при этом для каждой породы и породной группы рыб составляют генетический паспорт, представленный в таблице 1, в которой указано распределение 50-75 полиморфных микросателлитных маркеров, выявленных с помощью 5 пар праймеров (PR 4, 10, 19, 24, 26) по пяти соответствующим локусам (PRL 4, 10, 19, 24, 26), и по которой определяют породу рыб.
4. Способ по п.3, отличающийся тем, что размер амплифицируемых продуктов определяют с помощью маркера молекулярных масс (10 bp Ladder).
5. Способ по п.3, отличающийся тем, что набор контрольных амплификатов содержит образцы амплифицированной ДНК, характерные для каждой из изученных пород карповых рыб, в том числе карпа или сазана.
6. Способ по пп.3, 4 и 5, отличающийся тем, что верификацию породной принадлежности проверяют с помощью многомерного анализа, например путем дискриминантного анализа, метода главных компонент, в котором в качестве обучающей выборки служит выборка аллелей и аллельных сочетаний одной из набора пород рыб.
РИСУНКИ
MM4A – Досрочное прекращение действия патента СССР или патента Российской Федерации на изобретение из-за неуплаты в установленный срок пошлины за поддержание патента в силе
Дата прекращения действия патента: 26.03.2007
Извещение опубликовано: 10.07.2008 БИ: 19/2008
NF4A – Восстановление действия патента СССР или патента Российской Федерации на изобретение
Дата, с которой действие патента восстановлено: 27.04.2009
Извещение опубликовано: 27.04.2009 БИ: 12/2009
|
|